ggseg是2018年出的R工具包,可以在R中绘制到皮层或者皮层下区域的统计结果。和brainconn一样,解决了需要从R导出数据用其他软件作图的问题。ggseg基于ggplot,因此能用ggplot的方式调整作图效果,比如facet_wrap和theme,同时还兼容了dplyr的管道命令(%>%)。
在最近1.6的版本中做出了很多主要的更新,引入了geom_brain/geom_sf,更符合ggplot的规范,让作图的逻辑更加清晰。之前使用的ggseg()的作图方式仍然会保留一段时间,但是将来会被geom的方式取代。
一直在用1.5.3不想升级的人👇

软件目前自带两个常用模板:
1. aparc (也就是Desikan-Killany atlas)
2. aseg (Automatic subcortical segmentation)
当然还可以加载一些作者制作的模板。
ggseg容易上手,需要做的就是告诉软件每个region/label的数据是什么。本文介绍基本功,基于目前最新的版本1.6.02,详情参考软件的文章和vignettes。
Mowinckel, A. M., & Vidal-Piñeiro, D. (2020). Visualization of Brain Statistics With R Packages ggseg and ggseg3d. Advances in Methods and Practices in Psychological Science, 3(4), 466-483.

安装

install.packages("remotes")install_github("LCBC-UiO/ggseg", build_vignettes = TRUE)
⚠️github中有一个vignette有一些路径设置的问题,如果安装出错的话,build_vignettes选择FALSE。GitHub中的vignettes,但是不需要下载和knit,到软件主页查看作者织好的。

数据

比如需要绘制以下四个区域的统计值:
library(dplyr)
someData = tibble(
region = c("transverse temporal", "insula",
"precentral","superior parietal"),
p = sample(seq(0,.5,.001), 4)
)
这里用region作为匹配,需要region和模板中的名称一致,可以看到它不是缩写,有空格,而且没有left/right的信息。另外还可以使用label作为匹配,同样需要和模板中一致,label的形式更接近于实际使用中的情况。
someData = tibble(
label = c("lh_bankssts", "lh_insula",
"lh_parsopercularis","lh_parsorbitalis"),
p = sample(seq(0,.5,.001), 4)
)
下面的例子用region的数据,但实际使用中推荐label的方式。

作图

geom_brain() vs. geom_sf()
geom_brain和geom_sf用的是不同的风格,查看帮助文件会发现后者允许设置的内容更多,灵活度更高。
geom_brain
p1=ggplot(someData) +
geom_brain(atlas = dk,
position = position_brain(hemi ~ side),
aes(fill = p))p1
geom_sf
p1=someData %>%
brain_join(dk) %>%
reposition_brain(hemi ~ side) %>%
ggplot() +
geom_sf(aes(fill = p))p1
其中position_brain默认是水平,它设置是 rows ~ columns organisation。在geom_sf作图中需要使用reposition_brain的方式实现。
改theme
p1+theme_void()
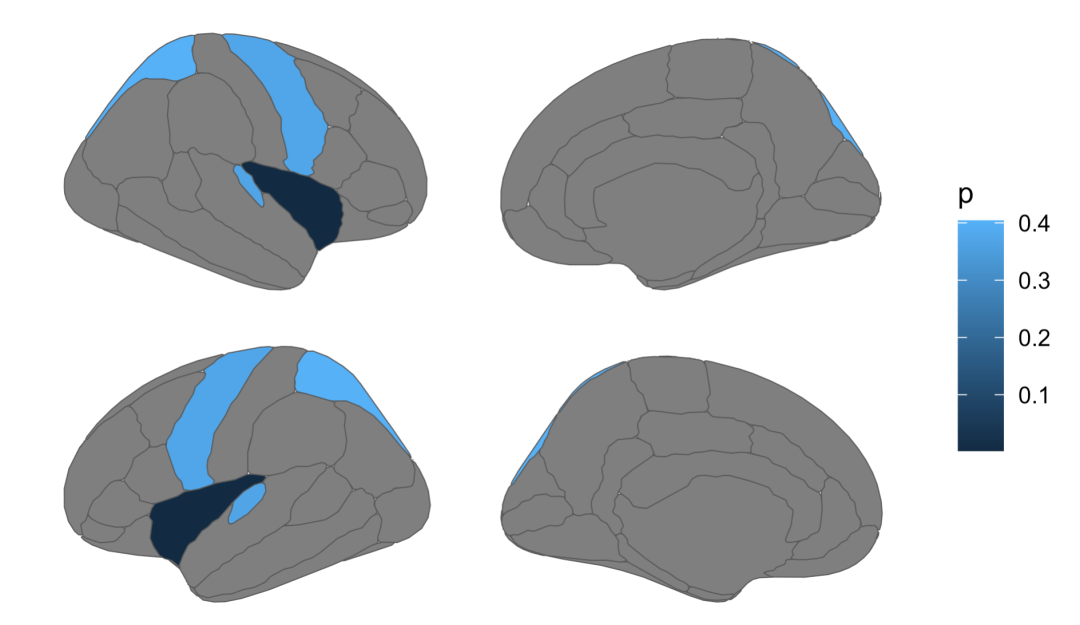
使用theme_void()/theme_brain2()是快速去掉坐标轴的方法,也可以使用theme_custombrain()/theme()做更多设置。⚠️ggseg用了mono字体,如果要拼图的话需要注意一些字体的统一性。
theme_custombrain(
plot.background = "white",
text.colour = "darkgrey",
text.size = 12,
text.family = "mono"
)
改colorbar
scale_fill_系列的函数都适用。
p1+scale_fill_gradient(low = "grey", high = "brown")+
theme_void()
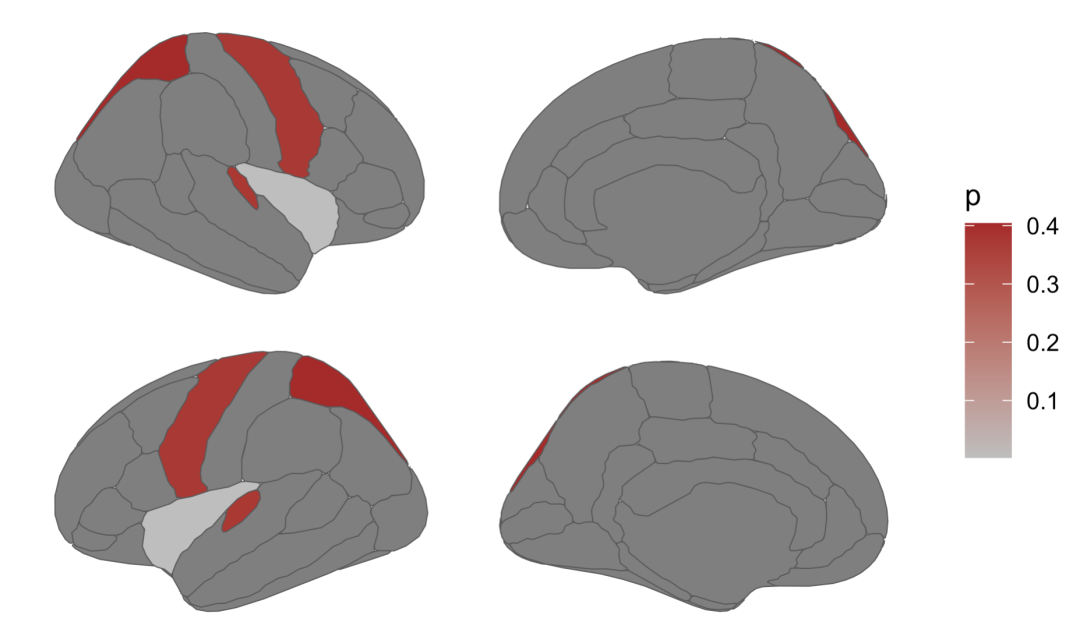
p1+scale_fill_distiller(palette = "RdPu")+
theme_void()
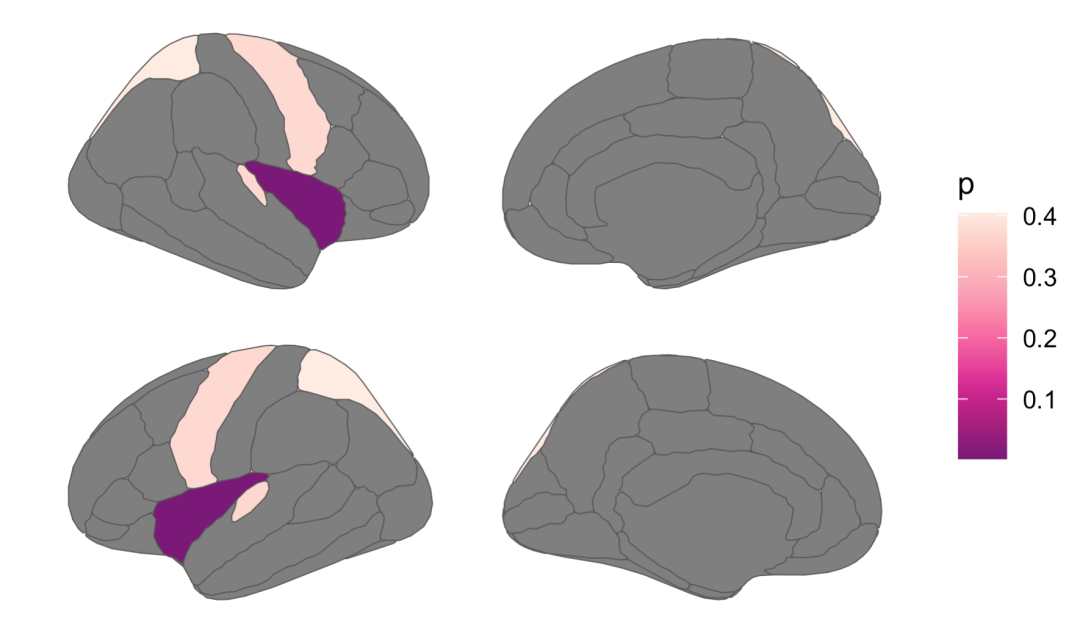
p1+scale_fill_viridis_c(option = "cividis", direction = -1)+
theme_void()
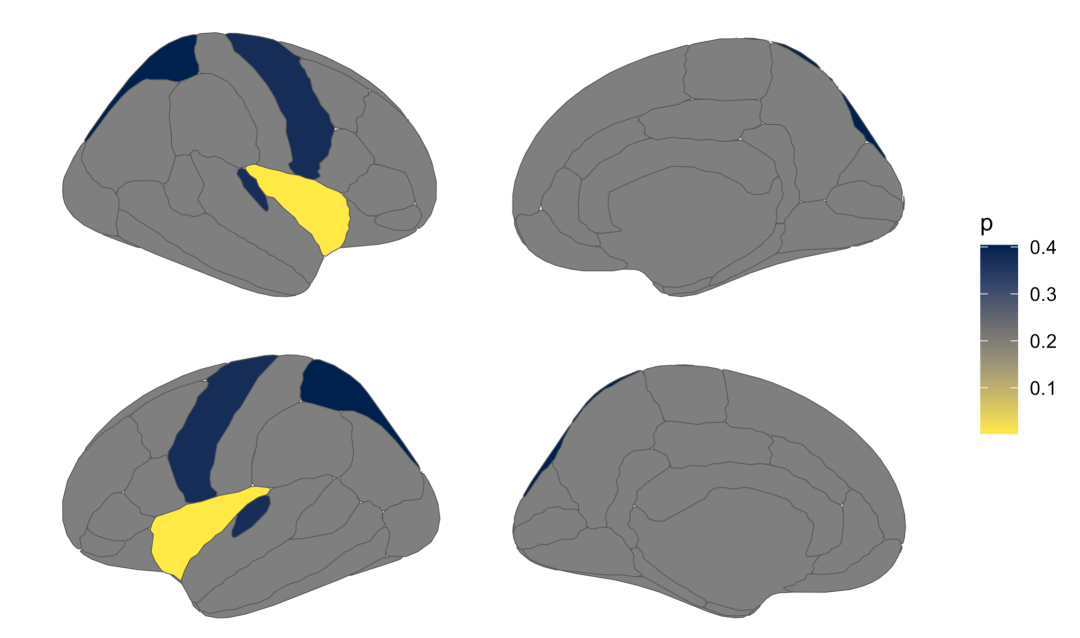
基本功能介绍到此结束,一些细节的修改比如增加title,修改legend的标题和位置,设置colorbar的范围等和ggplot的设置类似。对于subcortical volume的作图方法类似,同样是添加相应region/label的数据。最后,推荐用label+geom_sf的方式进行作图。

Extra


原理
dk其实是一堆数据,如果直接画dk中data会发现
plot(dk$data)
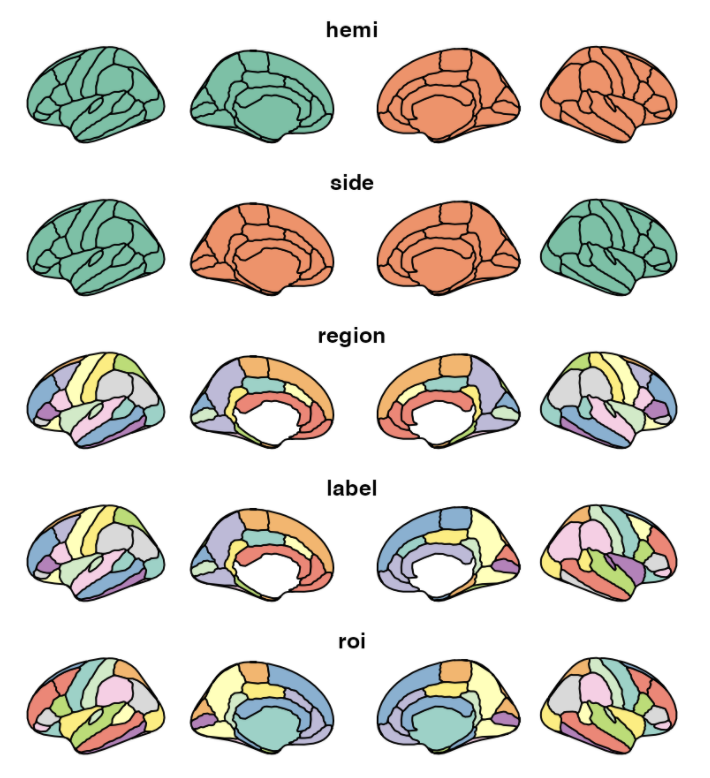
软件作图的逻辑其实是在dk的数据中增加一个新的column,作图时指定需要fill的column是哪个一个。
brain_join可以根据label或者region添加新column到dk中。
someData %>%
brain_join(dk)
当然也可以手动进行添加
dk %>%
as_tibble() %>%
left_join(someData)
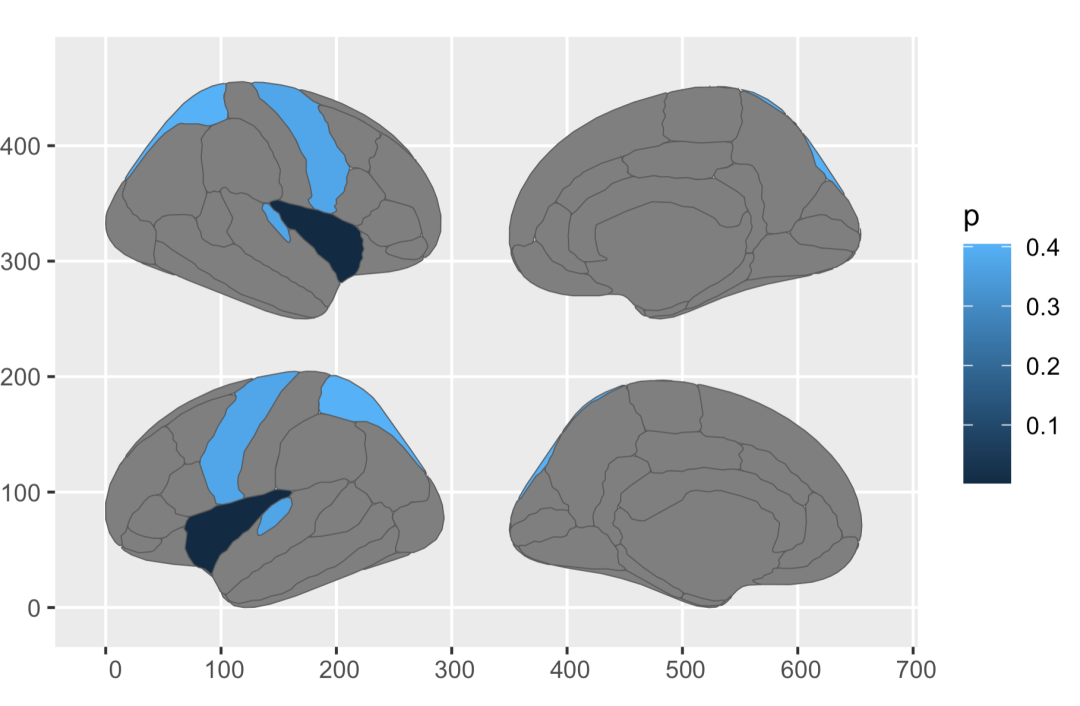
添加p值的column后如果不指定需要画哪一个column,得到下图
someData %>%
brain_join(dk) %>%
plot()
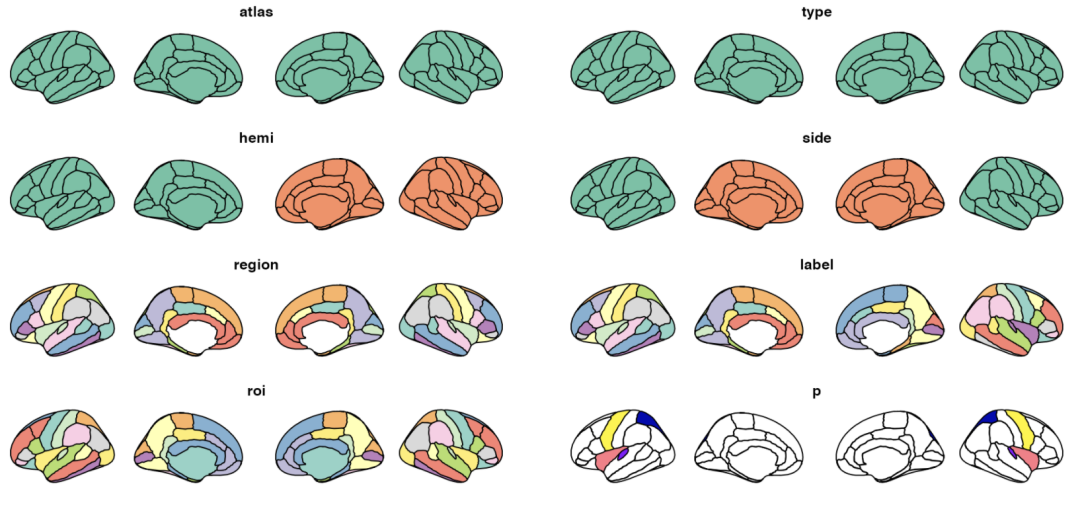
使用facet_wrap
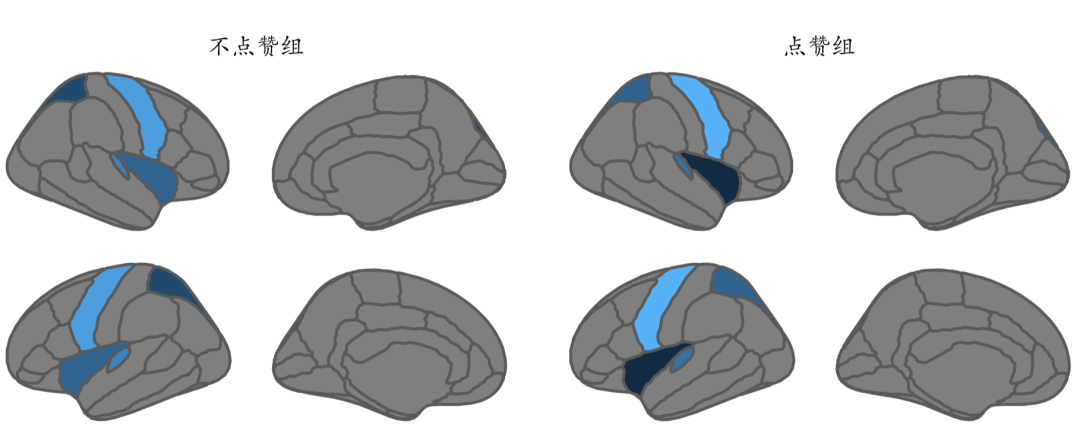
数据里增加分组的变量group,可以使用facet_wrap同时绘图。facet_wrap相比于先分组制图,后使用grid等工具拼接的方式,优点在于能用同一个colorbar。
someData <- tibble(
region = rep(c("transverse temporal", "insula",
"precentral","superior parietal"), 2),
p = sample(seq(0,.5,.001), 8),
groups = c(rep("点赞组", 4), rep("不点赞组", 4))
)
someData %>%
group_by(groups) %>%
brain_join(dk) %>%
reposition_brain(hemi ~ side) %>%
ggplot(aes(fill = p)) +
geom_sf(show.legend = FALSE) +
facet_wrap( ~ groups) +
theme_void()+
theme(text = element_text(family='Kai'))
读取freesurfer文件作图
作者提供了读取freesurfer统计结果文件的方式,可以一次读取统计文件中ROI的各种指标。"aparc.stats$"是正则表达式,说明lh/rh都会读取。
dat <- read_atlas_files(subject_dir, "aparc.stats$")
dat
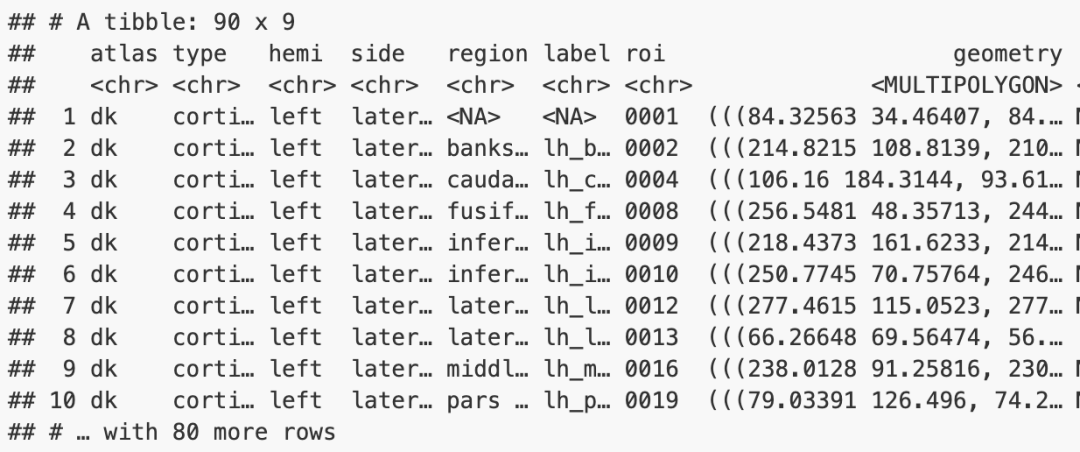
#> # A tibble: 68 x 11
#> subject label NumVert SurfArea GrayVol ThickAvg ThickStd MeanCurv GausCurv
#> <chr> <chr> <int> <int> <int> <dbl> <dbl> <dbl> <dbl>
#> 1 bert lh_bank… 1181 831 2297 2.77 0.428 0.116 0.024
#> 2 bert lh_caud… 843 572 1534 2.72 0.469 0.124 0.013
#> 3 bert lh_caud… 2758 1840 5772 2.80 0.526 0.114 0.021
#> 4 bert lh_cune… 2683 1654 3074 1.81 0.471 0.14 0.033
#> 5 bert lh_ento… 581 416 1840 3.33 0.667 0.116 0.032
#> 6 bert lh_fusi… 4113 2875 8519 2.72 0.599 0.131 0.025
#> 7 bert lh_infe… 4948 3466 10559 2.70 0.509 0.131 0.029
#> 8 bert lh_infe… 5056 3542 12358 2.98 0.625 0.138 0.033
#> 9 bert lh_isth… 1561 990 2350 2.09 0.745 0.113 0.023
#> 10 bert lh_late… 7961 5077 12743 2.30 0.588 0.139 0.033
#> # … with 58 more rows, and 2 more variables: FoldInd <int>, CurvInd <dbl>
画cortical thickness std
ggseg(dat, mapping = aes(fill = ThickStd))
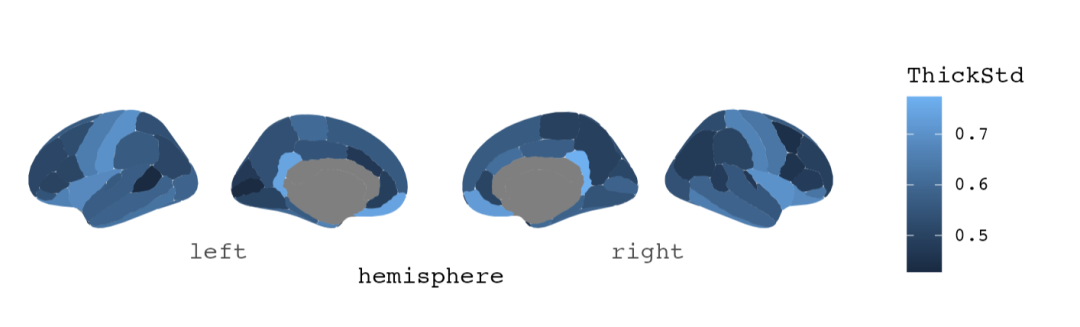
同时画几个指标。这只是功能展示,因为几个指标不在一个scale上。
dat %>%
gather(stat, val, -subject, -label) %>%
group_by(stat) %>%
ggseg(mapping = aes(fill = val)) +
facet_wrap(~stat)

实例
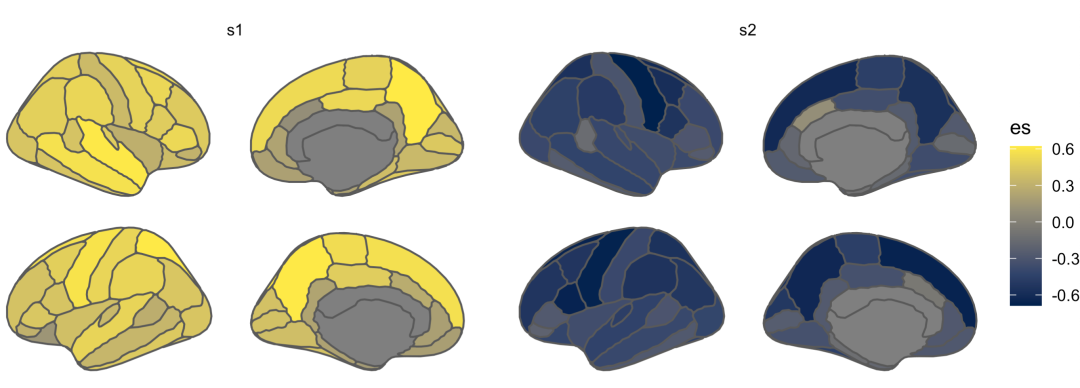
两个组s1和s2分别和正常组做了dk-68个区域皮层厚度的比较,结果如下:

制作effect size的统计图
#替换掉前缀后缀使得label和ggseg的一致
label=df$X
label=sub('combat_L','lh',label,ignore.case = TRUE)
label=sub('combat_R','rh',label,ignore.case = TRUE)
label=sub('_thickavg','',label,ignore.case = TRUE)
es=c(df$s1_es_vals,df$s2_es_vals)
#数据
someData = tibble(
label = rep(label,2),
es = es,
groups = c(rep("s1", 68), rep("s2", 68))
)
#作图
someData %>%
group_by(groups) %>%
brain_join(dk) %>%
reposition_brain(hemi ~ side) %>%
ggplot(aes(fill = es)) +
geom_sf(show.legend = TRUE) +
facet_wrap( ~ groups) +
scale_fill_viridis_c(option = "cividis")+
theme_void()