# 导入库import pandas as pd # panda库import matplotlib.pyplot as pltfrom sklearn.preprocessing import StandardScaler # 标准化库from sklearn.cluster import KMeans # 导入sklearn聚类模块from sklearn.metrics import silhouette_score # 效果评估模块from sklearn.decomposition import PCAimport matplotlib# 替换为你的文件路径data_path = r'E:\JetBrains\PythonProject\DeepLearning\provicedata.csv'df = pd.read_csv(data_path)# 选择特征列features = ['GDP', 'Primary industry', 'Primary industry','Primary industry','resident population','population growth rate','import and export','export value','import value','General budget revenue','General budget expenditure','Per capita disposable income','Per capita consumption expenditure','area'] # 替换为你的特征列名X = df[features]n_samples, n_features = X.shapeprint('样本数:%d , 特征数:%d' % (n_samples, n_features))# sklearn.cluster.KMeans(n_clusters=K)# 1. n_cluster:聚类个数(即K),默认值是8。# 2. init:初始化类中心的方法(即选择初始中心点的根据),默认“K-means++”,其他可选参数包括“random”。# 3. n_init:使用不同类中心运行的次数,默认值是10,即算法会初始化10次簇中心,然后返回最好的一次聚类结果。# 4. max_iter:单次运行KMeans算法的最大迭代次数,默认值是300。# 5. tol:浮点型,两次迭代之间簇内平方和下降的容忍阈值,默认为0.0001,如果两次迭代之间下降的值小于tol所设定的值,迭代就会停下。# 6. verbose:是否输出详细信息,参数类型为整型,默认值为0,1表示每隔一段时间打印一次日志信息。# 7. random_state:控制每次类中心随机初始化的随机种子,作用相当于能够锁定和复现同一次随机结果,默认为none,也可以随机设置数字。# 8. copy_x:在预先计算距离时,首先将数据居中在数值上更准确。如果 copy_x 为 True(默认),则不修改原始数据。# 如果为 False,则修改原始数据,并在函数返回之前放回,但通过减去再添加数据均值可能会引入小的数值差异。# 请注意,如果原始数据不是C-contiguous,即使copy_x 为False,也会进行复制。如果原始数据是稀疏的,但不是 CSR 格式,即使 copy_x 为 False,也会进行复制。# 9. algorithm:有三种参数可选:auto”, “full”, “elkan”,默认为auto。K-means 算法使用。# 经典的EM-style算法是“full”。通过使用三角不等式,“elkan” 变体对具有明确定义的集群的数据更有效。# 然而,由于分配了一个额外的形状数组(n_samples,n_clusters),它更加占用内存。# 数据归一化scaler = StandardScaler()scalerX = scaler.fit_transform(X)for i in range(2,5):# 1.components_ :特征空间中的主轴,表示数据中最大方差的方向。组件按排序 explained_variance_# 2.explained_variance_:它代表降维后的各主成分的方差值。方差值越大,则说明越是重要的主成分# 3.explained_variance_ratio_:它代表降维后的各主成分的方差值占总方差值的比例,这个比例越大,则越是重要的主成分# 4.singular_values_:每个特征的奇异值,奇异值等于n_components 低维空间中变量的2范数# 5.mean_:每个特征的均值# 6.n_components_:即是上面输入的参数值# 7.n_features_:训练数据中的特征数量# 8.n_samples_:训练数据中的样本数# 9.noise_variance_:等于X协方差矩阵的(min(n_features,n_samples)-n_components)个最小特征值的平均值pca = PCA(n_components=i)pcaX = pca.fit_transform(scalerX)print("For n_components={}, The PCA explained_variance_ratio_ is {}".format(i, pca.explained_variance_ratio_))# For n_components=2, The PCA explained_variance_ratio_ is [0.54113074 0.30908829]# For n_components=3, The PCA explained_variance_ratio_ is [0.54113074 0.30908829 0.06978044]# For n_components=4, The PCA explained_variance_ratio_ is [0.54113074 0.30908829 0.06978044 0.04070103]pca = PCA(n_components=2)pcaX = pca.fit_transform(scalerX)
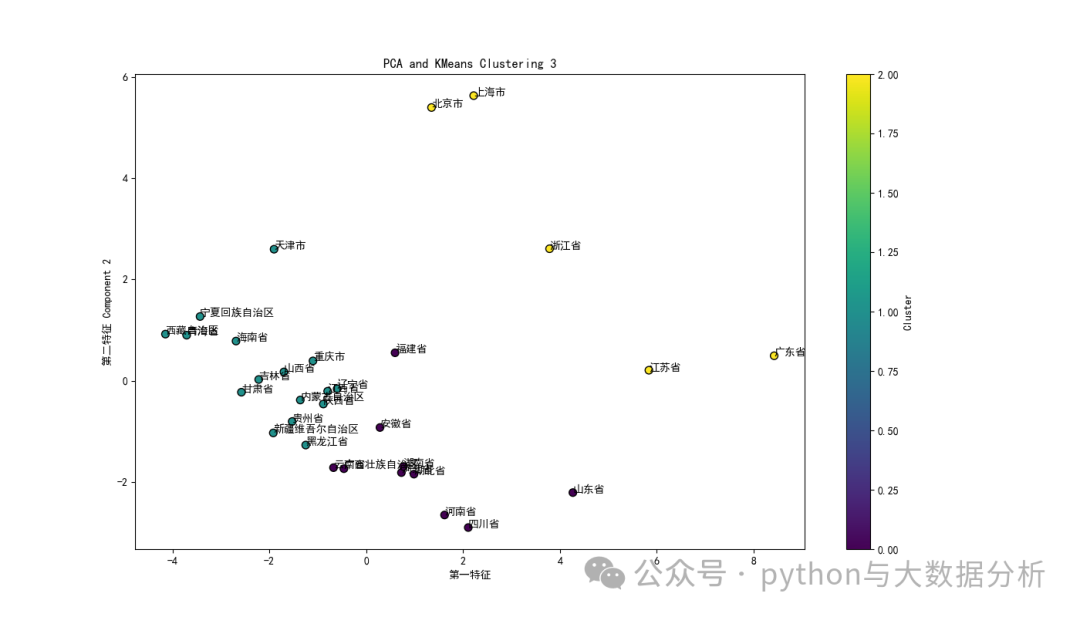
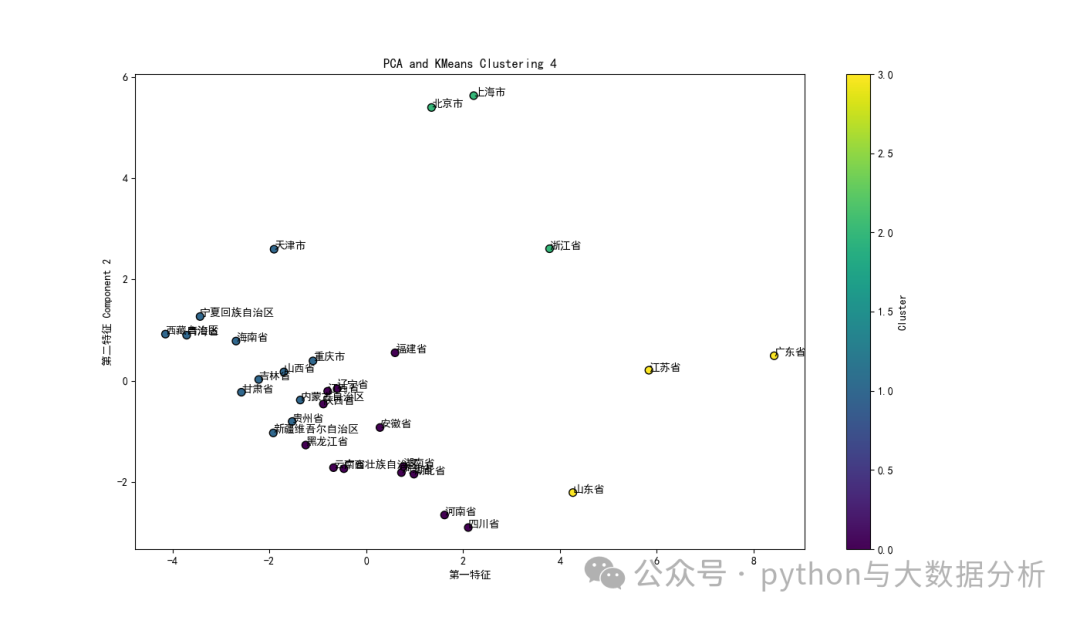
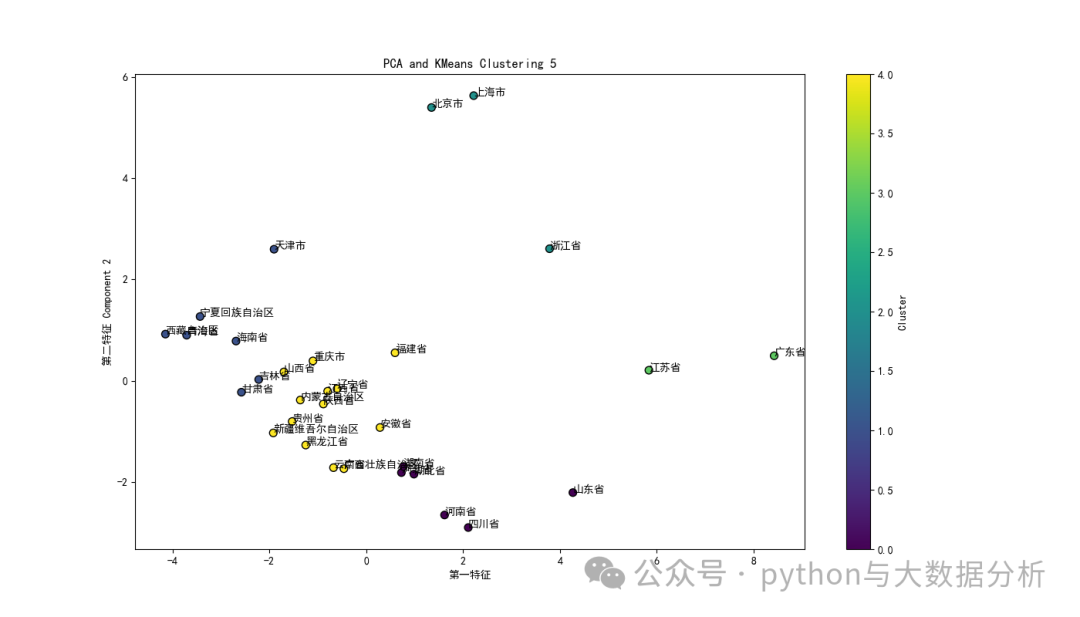
以下代码主要是在不同聚类类别聚类过程中,同时基于PCA的主成分进行特征可视化展示。
# 设置matplotlib正常显示中文和负号matplotlib.rcParams['font.family'] = 'SimHei' # 设置字体为黑体matplotlib.rcParams['axes.unicode_minus'] = False # 正确显示负号for k in range(3,6):# 模型训练KmeansModel = KMeans(n_clusters=k, random_state=42)KmeansModel.fit(pcaX)kmeansY = KmeansModel.predict(pcaX)# 计算轮轴系数:# 轮廓系数用于计算每个样本的平均簇内距离a(样本i到同簇其他样本的平均距离,ai值越小说明该样本越应该被聚到该类,即簇内不相似度)和平均邻近簇距离b(样本i到其他相邻簇的所有样本的平均距离bi,bi越大说明样本i越不属于其他簇,即簇间不相似度)。# 每个样本的轮廓系数计算公式为:(b-a)/Max(a,b),轮廓系数越接近1说明结果越好(聚类越准确),越接近-1说明结果越差,若值在0值附近,则说明样本在两个簇的边界上score = silhouette_score(pcaX, KmeansModel.labels_, metric='euclidean')print("For n_clusters={}, The Silhouette Coefficient is {}".format(k, score))# 合并数据和特征kmeans_labels = pd.DataFrame(KmeansModel.labels_, columns=['scaler pca kmean labels'+str(k)])df = df.join(kmeans_labels)# 绘制结果plt.figure(figsize=(14, 8))plt.scatter(pcaX[:, 0], pcaX[:, 1], c=KmeansModel.labels_, cmap='viridis', marker='o', edgecolor='k', s=50) # 绘制聚类结果for i, txt in enumerate(df['city']):plt.text(pcaX[:, 0][i], pcaX[:, 1][i], txt)plt.xlabel('第一特征')plt.ylabel('第二特征 Component 2')plt.title('PCA and KMeans Clustering '+str(k))plt.colorbar(label='Cluster') # 显示颜色条表示不同的聚类中心plt.show()
三个聚类簇时的可视化展现效果

文章转载自追梦IT人,如果涉嫌侵权,请发送邮件至:contact@modb.pro进行举报,并提供相关证据,一经查实,墨天轮将立刻删除相关内容。






