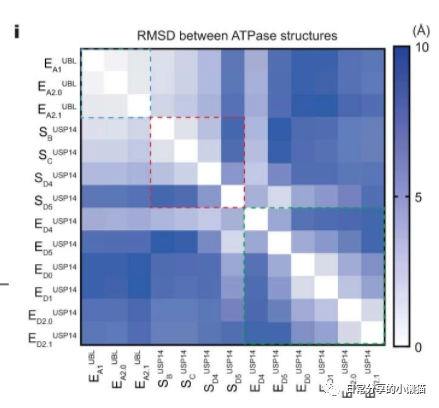
本文主要展示如何利用R语言绘制来自nature期刊USP14-regulated allostery of the human proteasome by time-resolved cryo-EM(Shuwen Zhang, et al,2022)[1]一文中的Extended Data Fig.7i。该图形式为热图。如下图所示:

1、图形绘制
setwd("C:\\Users\\Acer\\Desktop")
library(dplyr)
library(ggplot2)
zhang_ed.fig7.i <- readxl::read_xlsx("41586_2022_4671_MOESM7_ESM.xlsx", sheet = 2)
head(zhang_ed.fig7.i)
# type EA1UBL EA2.0UBL EA2.1UBL SBUSP14 SCUSP14 SD4USP14 SD5USP14 ED4USP14 ED5USP14 ED0USP14 ED1USP14 ED2.0USP14 ED2.1USP14
# <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#1 EA1UBL 0 0.388 0.75 1.29 2.06 3.33 6.78 3.96 7.16 8.04 8.02 6.99 6.76
#2 EA2.0UBL 0.388 0 0.708 1.34 2.08 3.34 6.86 3.91 7.25 8.00 7.92 7.07 6.70
#3 EA2.1UBL 0.75 0.708 0 1.95 2.62 3.72 6.98 4.04 7.27 8.28 8.32 7.63 7.29
#4 SBUSP14 1.29 1.34 1.95 0 1.14 2.95 7.72 3.54 8.39 7.40 7.24 6.58 6.49
#5 SCUSP14 2.06 2.08 2.62 1.14 0 2.20 7.38 3.10 8.15 7.26 7.12 6.56 6.41
#6 SD4USP14 3.33 3.34 3.72 2.95 2.20 0 5.12 2.56 5.8 7.19 7.20 6.78 6.87
fig7.i <- zhang_ed.fig7.i %>% reshape2::melt(id.vars = "type", variable.name = "class", value.name = "value")
fig7.i$type <- factor(fig7.i$type, levels = fig7.i$type %>% unique())
fig7.i$class <- factor(fig7.i$class, levels = fig7.i$class %>% unique() %>% rev() )
fig7.i <- fig7.i %>% mutate(integer.x = as.numeric(fig7.i$type)) #factor to numeric
fig7.i <- fig7.i %>% mutate(integer.y = as.numeric(fig7.i$class)) #factor to numeric
head(fig7.i)
# type class value integer.x integer.y
#1 EA1UBL EA1UBL 0.000 1 13
#2 EA2.0UBL EA1UBL 0.388 2 13
#3 EA2.1UBL EA1UBL 0.750 3 13
#4 SBUSP14 EA1UBL 1.288 4 13
#5 SCUSP14 EA1UBL 2.058 5 13
#6 SD4USP14 EA1UBL 3.331 6 13
str(fig7.i)
#'data.frame': 169 obs. of 5 variables:
# $ type : Factor w/ 13 levels "EA1UBL","EA2.0UBL",..: 1 2 3 4 5 6 7 8 9 10 ...
# $ class : Factor w/ 13 levels "ED2.1USP14","ED2.0USP14",..: 13 13 13 13 13 13 13 13 13 13 ...
# $ value : num 0 0.388 0.75 1.288 2.058 ...
# $ integer.x: num 1 2 3 4 5 6 7 8 9 10 ...
# $ integer.y: num 13 13 13 13 13 13 13 13 13 13 ...
#绘图
ggplot(fig7.i, aes(x = type, y = class, fill = value)) +
geom_tile() +
scale_x_discrete(expand = c(0,0)) +
scale_y_discrete(expand = c(0,0)) +
geom_rect(aes(xmin=1 - 0.45, xmax = 3 + 0.45, ymin = 11 - 0.45, ymax = 13 + 0.45), color = "red", fill = NA, size = 1, linetype = 2) +
geom_rect(aes(xmin=4 - 0.45, xmax = 7 + 0.45, ymin = 7 - 0.45, ymax = 10 + 0.45), color = "blue", fill = NA, size = 1, linetype = 2) +
geom_rect(aes(xmin=8 - 0.45, xmax = 13 + 0.45, ymin = 1 - 0.45, ymax = 6 + 0.45), color = "green", fill = NA, size = 1, linetype = 2) +
scale_fill_distiller(palette = "Purples", direction = 1, breaks = seq(0,10,5), limits = c(0,10)) +
theme_bw() +
theme(axis.text.x = element_text(angle = 90, hjust = 1),
text = element_text(colour = "black"),
plot.title = element_text(hjust = 0.5),
axis.text = element_text(size = 10),
axis.title = element_blank(),
axis.ticks.length = unit(0, "cm")) +
guides(fill = guide_colorbar(barheight = 22, frame.colour = "black", ticks.colour = "black")) +
labs(fill = " (A)", title = "RMSD between ATPase structures")
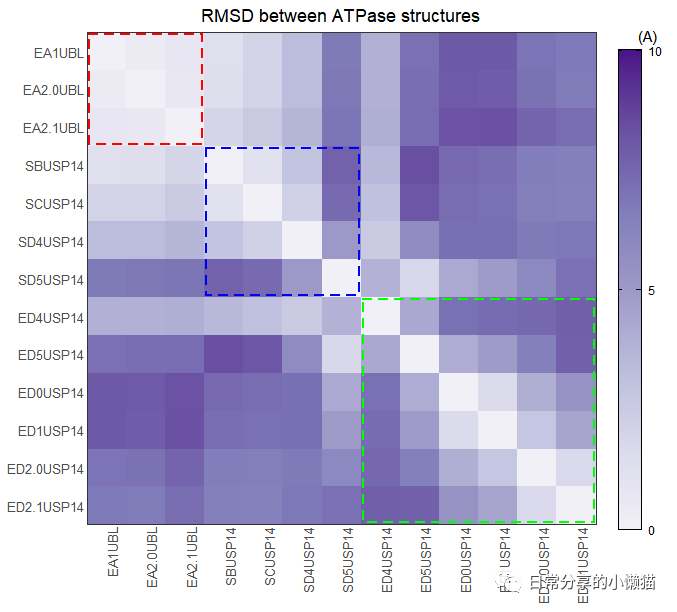
该图所使用的基本绘图函数为geom_tile() 函数与geom_rect() 函数。其中有2个需要注意的点:一是如何在热图中加入框线,为此首先需要将因子型变量转换为数值型变量,即用到as.numeric() 函数,其次根据因子的对应数值,填充到geom_rect() 函数中,来完成框线的绘制;二是因子的顺序,需要对其中一列因子顺序进行反转。
2、其他
其他绘图方法可进一步阅读公众号其他文章。
如有帮助请多多点赞哦!
参考资料
Zhang, S., Zou, S., Yin, D. et al. USP14-regulated allostery of the human proteasome by time-resolved cryo-EM. Nature 605, 567–574 (2022). : https://doi.org/10.1038/s41586-022-04671-8
文章转载自日常分享的小懒猫,如果涉嫌侵权,请发送邮件至:contact@modb.pro进行举报,并提供相关证据,一经查实,墨天轮将立刻删除相关内容。






