之前看到大佬写的infercnvpy的帖子用python版InferCNVpy加速运算,然后发现目前好像没有从seurat对象转到infercnvpy的教程,所以写一下我的操作方式,比较傻瓜~
首先是安装需要的包,这里我直接使用生信技能树的教程。
BiocManager::install("GEOquery")
url='https://gitee.com/jmzeng/annoprobe.git'
library(remotes)
install_git(url)
library(AnnoProbe)
library(Seurat)
library(infercnv)
然后载入seurat对象
cells.use=colnames(sc)[which(sc$CellType %in% c("Epithelial_cells","B_cells","Oligo&Astrocytes"))]dat=as.data.frame(GetAssayData(subset(sc, cells=cells.use)))Idents(sc)<-"CellType"groupinfo=data.frame(v1=colnames(dat),v2=sc@active.ident[cells.use])
geneInfor=annoGene(rownames(dat),"SYMBOL",'human')colnames(geneInfor)geneInfor=geneInfor[with(geneInfor, order(chr, start)),c(1,4:6)]geneInfor=geneInfor[!duplicated(geneInfor[,1]),]length(unique(geneInfor[,1]))head(geneInfor)dat=dat[rownames(dat) %in% geneInfor[,1],]dat=dat[match(geneInfor[,1], rownames(dat) ),]dim(dat)groupFiles='groupFiles.txt'head(groupinfo)write.table(groupinfo,file = groupFiles,sep = '\t',quote = F,col.names = F,row.names = F)expFile='expFile_t.txt'## 注意这里的exp和R版不一样,做了转置write.table(t(dat),file = expFile,sep = '\t',quote = F)geneFile='geneFile.txt'write.table(geneInfor,file = geneFile,sep = '\t',quote = F,col.names = F,row.names = F)
到此就完成了R部分,下面切换到python。
安装教程参考大佬们的帖子,conda一般不会有问题。
import scanpy as scimport infercnvpy as cnvimport pandas as pdadata = sc.read_text('expFile_t.txt')anno = pd.read_table('groupFiles.txt',index_col='Id') ## 这里要对文件做相应更改,添加一个列名adata.obs['cell_groups'] = anno['cells']gene = pd.read_table('geneFile.txt',index_col='Gene') ## 同样做相应更改,添加列名# "chromosome", "start", "end"adata.var=gene


# 后面教程就差不多了
cnv.tl.infercnv(
adata,
reference_key="cell_groups",
reference_cat=[
"B_cells",
"Oligo&Astrocytes",
],
window_size=250,
)
cnv.pl.chromosome_heatmap(adata, groupby="cell_groups")
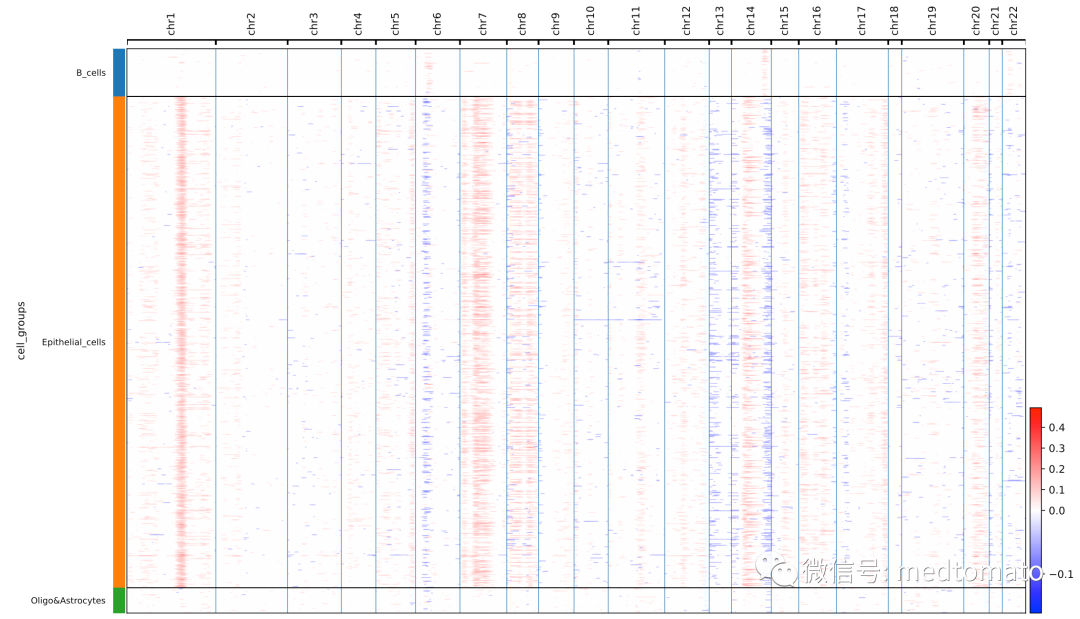
速度很快,接近2w个细胞也就一分钟左右吧。但是infercnvpy只是方法学相同,有朋友说和R版得到的结果还是有区别,使用需谨慎。
This package is still experimental. The results have not been validated, except in that they look similar, but not identical, to the results of InferCNV.

希望在一线的老师和朋友们都保重身体!尽快康复!大家在平时生活中也做好防护,熬过最艰难的时刻,一定会好的!
文章转载自西红柿的空间转录组,如果涉嫌侵权,请发送邮件至:contact@modb.pro进行举报,并提供相关证据,一经查实,墨天轮将立刻删除相关内容。






