❝一句话简介:一个可以用于探索、下载和简单分析 UCSC Xena data hubs 上所有数据集的 R Shiny 交互式应用。
❞
项目地址:https://github.com/openbiox/UCSCXenaShiny[1]
可以单独作为 R 包下载和使用,目前主要开发了数据集的下载和单基因的分析功能,很多都还需要完善和增加,欢迎在 https://github.com/openbiox/UCSCXenaShiny/issues[2] 提供反馈和建议。
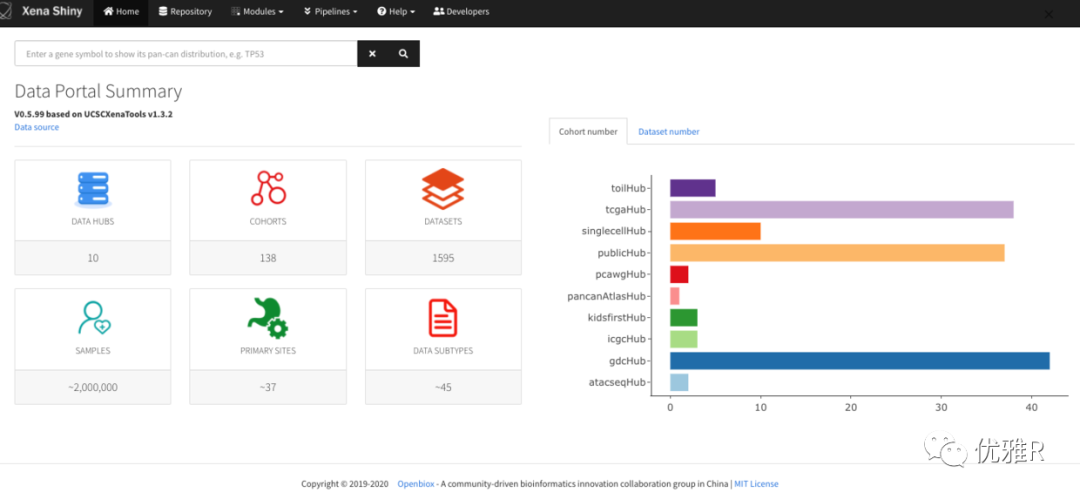
主界面:
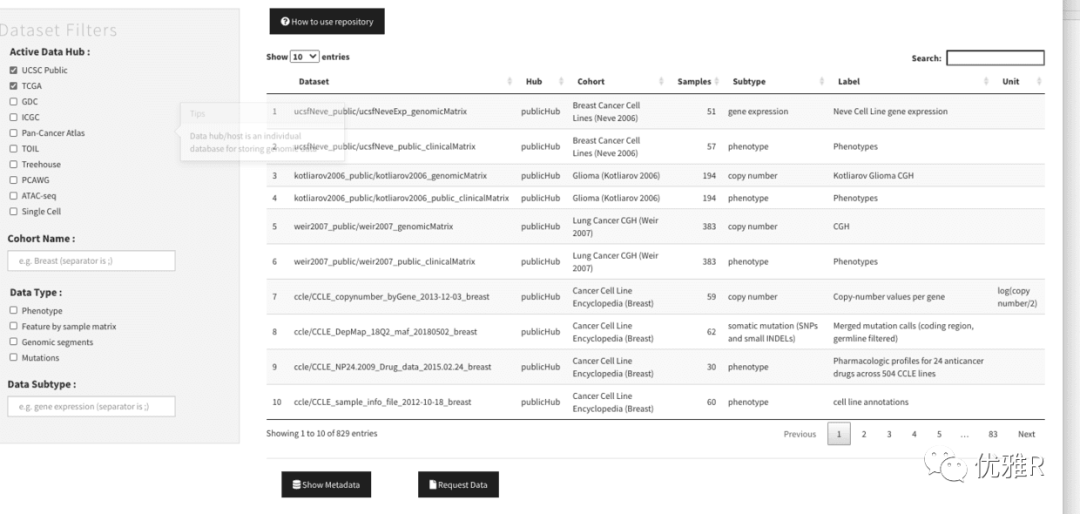
数据集的选择、查看和下载:
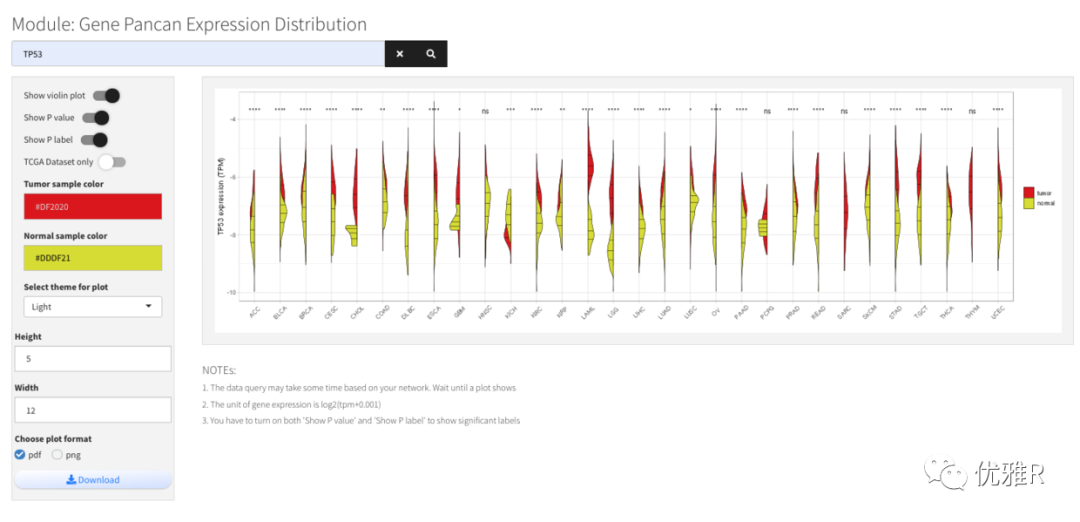
一些单基因分析模块:包括泛癌表达、生存分析、Cox分析等
接着看下目前6位参与的开发人员,如果没有他们就没有这个工具的存在啦。

该工具是 Openbiox 社区的项目之一,最近 Openbiox 社区推出了便捷的可视化和科研分析平台 Hiplot(知乎有介绍文章 https://zhuanlan.zhihu.com/p/161098733[3]),UCSCXenaShiny 作为高级功能模块之一。
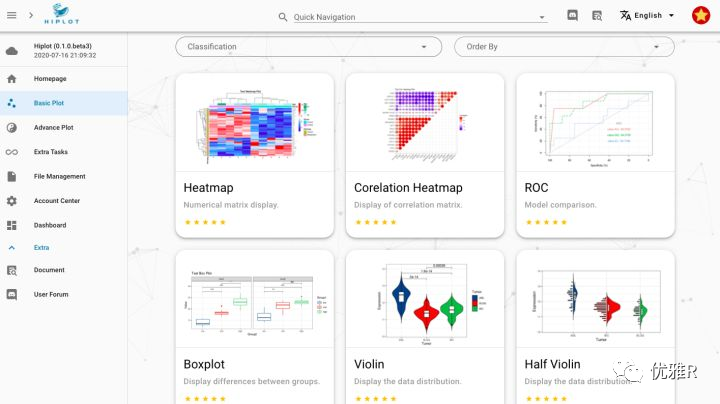
目前该平台正在内测,如果你不想要安装 R 包,又想要尝试一下 UCSCXenaShiny,欢迎注册

最后,如果这个工具能够帮助到你的科研工作,记得引用一下我们的预印本:
❝Wang, S.; Xiong, Y.; Gu, K.; Zhao, L.; Li, Y.; Zhao, F.; Li, X.; Liu, X. UCSCXenaShiny: An R Package for Exploring and Analyzing UCSC Xena Public Datasets in Web Browser. Preprints 2020, 2020070179 (doi: 10.20944/preprints202007.0179.v1).
❞
Reference
https://github.com/openbiox/UCSCXenaShiny: https://github.com/openbiox/UCSCXenaShiny
[2]https://github.com/openbiox/UCSCXenaShiny/issues: https://github.com/openbiox/UCSCXenaShiny/issues
[3]https://zhuanlan.zhihu.com/p/161098733: https://zhuanlan.zhihu.com/p/161098733






