| 了解了Chipseq的测试原理,这一章节将继续概述Chipseq常规分析点和使用的软件 |
Chipseq实验步骤:生信下机数据基础知识(8)-Chipseq概述
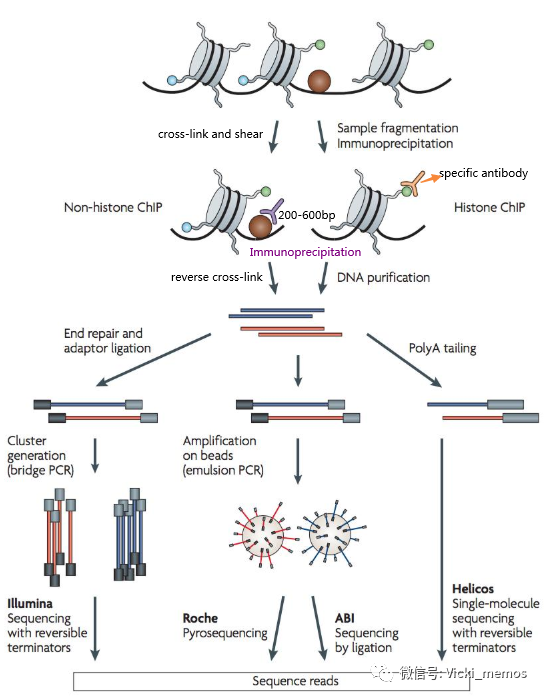
补充步骤-上述图中,在得到了纯化的DNA后,片段可以通过三种测序方式进行测序分析[1]:
1)(用的比较多的)使用bridge PCR富集目的DNA片段,然后Illumina测序分析(边合成边测序),一般为100bpPE测序策略或者150bpPE测序策略;2)使用emulsion PCR富集目的DNA片段,然后使用Roche454或者ABI测序平台测序(较早的方法);3)单分子测序平台,使用荧光核苷酸插入模板,这样子就可以便于摄像,而不需要额外的克隆,扩增步骤;
Chipseq的数据结果好坏直接由诸多因素影响,包括:样品量,样本处理,测序质量以及基因组复杂度等因素。
| 初次接触Chipseq项目的小白需要注意到,Chipseq分析中比较重要的一点就是阳性对照组的存在,也就是我们常说的input。input对照组是啥呢?是指上述实验过程中,断裂后的DNA片段,不经过免疫沉淀,直接经过逆转录交联,DNA纯化,以及随后的PCR等其他检测方法,获取到的DNA基因内碎片。由于input数据是完整的基因组被随机打断后,未经过选择的片段,所以有可能可以看到任何片段,只是由于没有进行特异富集,所以input对照实验组对应位置的data就会低很多。通过使用input对照获取到的实验组的peak峰值可以在一定水平上排除基础水平表达高的或者一些非特异性结合造成的假阳性peaks。因此,在后期分析的时候,input数据对于进行特定蛋白结合的DNA region的峰值是很有必要的。1 测定随机剪短的片段的效果如何,目标蛋白结合到region在样品中的表达情况;2 使用input对照获取到的实验组的peak峰值可以在一定水平上排除基础水平表达高的或者一些非特异性结合造成的假阳性peaks,相当于使用input数据来对样本数据进行归一化处理,去除背景噪音处理等。 |
总结一下Chipseq作用:
1 对所测物种,在全基因组范围内获取转录因子或者组蛋白修饰的DNA靶点
2 定义转录因子结合位点
3 将RNA测序,ATACseq和甲基化分析结合起来,揭示了基因转录前调控,转录调控和转录后调控机制以及网络调控图
4 获取所测物种全基因组范围内特定结合doman的谱系图
5 结合上述直接作用,可以用来对疾病病理机制,细胞分化机制,染色质动态图谱等进行深入的综合研究
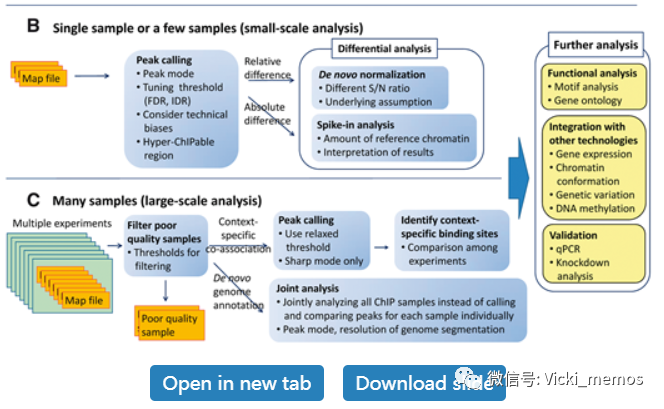
上图中展示的是测序数据经过初步数据过滤,以及比对后,后续的详细的分析点。比对软件使用常规的bwa或者bowtie2即可。
常规的分析:
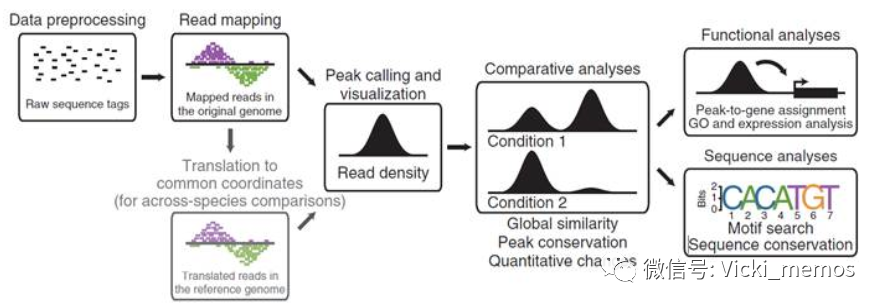
3 call peak分析,获取具有峰值reads的genome region区间以及峰值;4 可视化,可以使用igv或者UCSC的browser;
6 功能分析结合TF数据,筛选目的region,以及功能网络图,例如GO,KEGG等;
ps:其实Chipseq分析和ATACseq分析有些类似,可以相互参照;进一步的高级分析部分可以参照参考文献【2】根据需要了解,后期如果有需要也会概述一下;
使用的常规软件:
1.FastQC/Fxtools
2.Bowtie1/2/BWA
3.SAMtools+bedtools+ChIPQC
4.UCSC Tools/IGV
5.MACS2/MACS/ChIPseqR/Chipseq
6.deepTools/meme suite/ChIPpeakAnno/Homer/
ps:常规分析目前网上还有很多现成的工具可以使用,也有R包,python包可以使用,并不局限与上述的基础分析软件。
[1]Park, P. ChIP–seq: advantages and challenges of a maturing technology. Nat Rev Genet 10, 669–680 (2009). https://doi.org/10.1038/nrg2641
[2]Ryuichiro Nakato, Katsuhiko Shirahige.ecent advances in ChIP-seq analysis: from quality management to whole-genome annotation .Briefings in Bioinformatics, Volume 18, Issue 2, March 2017, Pages 279–290, https://doi.org/10.1093/bib/bbw023


往期回顾:
生信下机数据基础知识(9)-Chipseq概述二
生信下机数据基础知识(8)-Chipseq概述
生信下机数据基础知识(7)
生信下机数据基础知识(6)











