Bio如何撑起Redis后台半壁江山
干货:
Bio设计模式-----生产者消费者模式 Aof异步刷盘-----Bio关键先生 Bio引入Lazy free-----大Key删除
Redis引入Bio这套设计可以说支撑起了异步后台任务处理的半壁江山,为什么这么说呢?我们先来看下redis阻塞点:
集合全量查询和聚合操作; bigkey 删除; 清空数据库; AOF 日志同步写; 从库加载 RDB 文件
适合后台能处理的就有三个,2,3,4都适合后台子线程去处理,而且随着redis的发展,可能需要后台处理的越来越多,如何设计优秀的架构模式来支持现有和将来的需要异步处理的任务呢?答案就是:Bio系统 基于锁和共享变量来实现多线程生产者消费者模型
。
1. BIO系统设计与实现
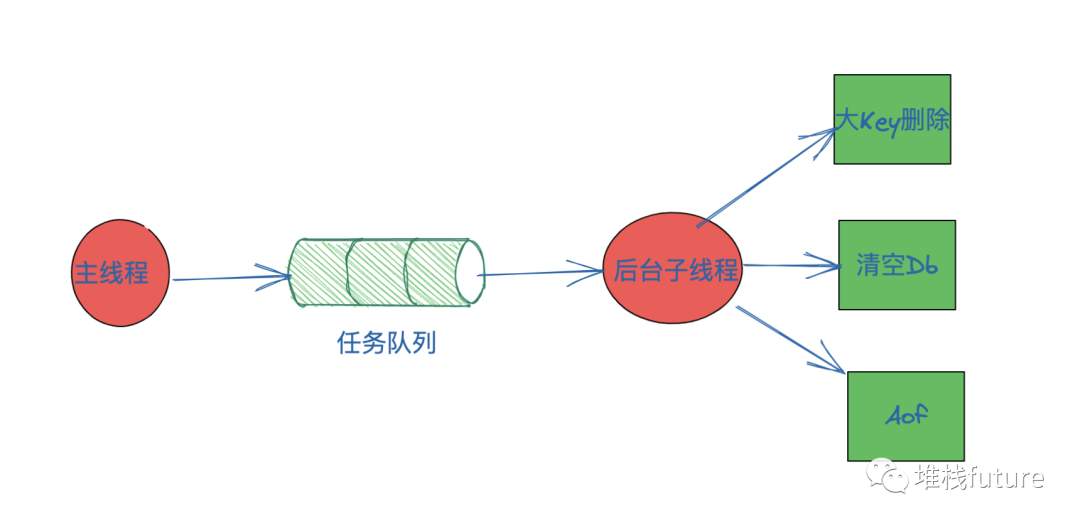
1.1 流程图
1.2 流程介绍
Redis 主线程启动后,会使用操作系统提供的 pthread_create 函数创建 3 个子线程,分别 由它们负责 AOF 日志写操作、键值对删除以及文件关闭的异步执行。主线程通过一个链表形式的任务队列和子线程进行交互。当收到键值对删除和清空数据库 的操作时,主线程会把这个操作封装成一个任务,放入到任务队列中,然后给客户端返回 一个完成信息,表明删除已经完成。但实际上,这个时候删除还没有执行,等到后台子线程从任务队列中读取任务后,才开始 实际删除键值对,并释放相应的内存空间。因此,我们把这种异步删除也称为惰性删除 (lazy free)。此时,删除或清空操作不会阻塞主线程,这就避免了对主线程的性能影 响。和惰性删除类似,当 AOF 日志配置成 everysec 选项后,主线程会把 AOF 写日志操作封 装成一个任务,也放到任务队列中。后台子线程读取任务后,开始自行写入 AOF 日志,这 样主线程就不用一直等待 AOF 日志写完了。
这里有个地方需要你注意一下,异步的键值对删除和数据库清空操作是 Redis 4.0 后提供 的功能
,Redis 也提供了新的命令来执行这两个操作
。
键值对删除:当你的集合类型中有大量元素(例如有百万级别或千万级别元素)需要删除时,我建议你使用 UNLINK 命令
。清空数据库:可以在 FLUSHDB 和 FLUSHALL 命令后加上 ASYNC 选项,这样就可以让后台子线程异步地清空数据库,如下所示:
FLUSHDB ASYNC
FLUSHALL AYSNC复制
2. 源码分析更清楚
因为涉及到的源码还算可以,所以想黏贴出来让大家直接看,加油哇。
1. 任务的创建和初始化
对于一个任务,比如aof持久化任务,首先要初始化一个队列,在redis里面使用了redis本身的链表结构创建这个队列。这个队列须要满足如下特点:
生产者发送任务到队列中。 若是队列不为空,消费者从队列中取任务;不然消费者进入等待状态。
这里的消费者就是后台线程,而为了完成队列为空则等待的功能,redis使用了条件变量机制,其初始化代码以下:
//后台线程数组 大小是BIO_NUM_OPS 在redis的3.2.3版本中是常量2, 现在版本中是BIO_NUM_OPS=3
//#define BIO_NUM_OPS 3
//表示支持三种任务。对于每种任务,对应一个list用于存储任务
static pthread_t bio_threads[BIO_NUM_OPS];
//bio_mutex和bio_condvar用于控制并发
static pthread_mutex_t bio_mutex[BIO_NUM_OPS];
static pthread_cond_t bio_newjob_cond[BIO_NUM_OPS];
static pthread_cond_t bio_step_cond[BIO_NUM_OPS];
//存储任务
static list *bio_jobs[BIO_NUM_OPS];
//存储pending状态的任务数量
static unsigned long long bio_pending[BIO_NUM_OPS];复制
创建好变量之后就开始初始化bioInit
初始化函数bioInit
//初始化锁与条件变量
for (j = 0; j < BIO_NUM_OPS; j++) {
pthread_mutex_init(&bio_mutex[j],NULL);
pthread_cond_init(&bio_newjob_cond[j],NULL);
pthread_cond_init(&bio_step_cond[j],NULL);
bio_jobs[j] = listCreate();
bio_pending[j] = 0;
}
......
......
//初始化线程 生成制定BIO_NUM_OPS数量的线程 线程传入一个编号j,0表明关闭文件,1表明aof初始化,2就是删除大Key
for (j = 0; j < BIO_NUM_OPS; j++) {
void *arg = (void*)(unsigned long) j;
if (pthread_create(&thread,&attr,bioProcessBackgroundJobs,arg) != 0) {
serverLog(LL_WARNING,"Fatal: Can't initialize Background Jobs.");
exit(1);
}
bio_threads[j] = thread;
}复制
在完成初始化任务之后,Redis就有了BIO_NUM_OPS个链表来表示任务队列,有BIO_NUM_OPS个线程调用bioProcessBackgroundJobs函数进行任务处理,参数是一个编号j,而且每一个队列都初始化了锁与条件变量作并发控制。
2. 任务入队列
任务入队列就是把一个任务放到链表的头部,而且把相应任务的pending值+1,表示这个队列里面未完成的任务多了一个。其中任务的结构如下:
struct bio_job {
//job任务结构
int fd; /* Fd for file based background jobs */
lazy_free_fn *free_fn; /* Function that will free the provided arguments */
void *free_args[]; /* List of arguments to be passed to the free function */
};复制
//提交Aof任务到队列
void bioCreateFsyncJob(int fd) {
//申请任务job空间
struct bio_job *job = zmalloc(sizeof(*job));
job->fd = fd;
//提交任务到队列
bioSubmitJob(BIO_AOF_FSYNC, job);
}
//提交Lazy free任务到队列
void bioCreateLazyFreeJob(lazy_free_fn free_fn, int arg_count, ...) {
va_list valist;
/* Allocate memory for the job structure and all required
* arguments */
struct bio_job *job = zmalloc(sizeof(*job) + sizeof(void *) * (arg_count));
job->free_fn = free_fn;
va_start(valist, arg_count);
for (int i = 0; i < arg_count; i++) {
job->free_args[i] = va_arg(valist, void *);
}
va_end(valist);
bioSubmitJob(BIO_LAZY_FREE, job);
}
//提交关闭文件任务到队列
void bioCreateCloseJob(int fd) {
struct bio_job *job = zmalloc(sizeof(*job));
job->fd = fd;
bioSubmitJob(BIO_CLOSE_FILE, job);
}
//提交任务
void bioSubmitJob(int type, struct bio_job *job) {
pthread_mutex_lock(&bio_mutex[type]);
listAddNodeTail(bio_jobs[type],job);
bio_pending[type]++;
pthread_cond_signal(&bio_newjob_cond[type]);
pthread_mutex_unlock(&bio_mutex[type]);
}
//返回指定类型的peding状态的作业的数量
unsigned long long bioPendingJobsOfType(int type) {
unsigned long long val;
pthread_mutex_lock(&bio_mutex[type]);
val = bio_pending[type];
pthread_mutex_unlock(&bio_mutex[type]);
return val;
}复制
先拿Aof入队列作为事例讲解:先调用bioCreateFsyncJob为任务结构分配空间,而后调用bioSubmitJob函数使用listAddNodeTail函数把任务放到链表的头部。这里使用的是redis本身实现的链表。能够看到,进行链表操做的时候,要先加锁,这是由于这里的链表是共享资源。在任务成功加入队列之后,调用pthread_cond_signal函数,通知阻塞等待的线程继续执行。上面这个过程是共享变量使用的基本模式: 加锁、置条件为真(这里是任务入队列)、通知、解锁
。
3. 任务出队列
Redis已经作好了任务初始化的工做,而且能够在队列里面放置新的任务,那么当队列里面有任务的时候,第一步初始化的时候开启的后台线程就会调用bioProcessBackgroundJobs函数进行出队处理任务,其处理主要代码如下:
void *bioProcessBackgroundJobs(void *arg) {
struct bio_job *job;
unsigned long type = (unsigned long) arg;
//一堆校验 省略
。。。。。。
//加锁
pthread_mutex_lock(&bio_mutex[type]);
while(1) {
listNode *ln;
/* The loop always starts with the lock hold. */
if (listLength(bio_jobs[type]) == 0) {
//条件不成立,等待
pthread_cond_wait(&bio_newjob_cond[type],&bio_mutex[type]);
//被通知之后,中止阻塞,从新判断条件
continue;
}
/* Pop the job from the queue. */
ln = listFirst(bio_jobs[type]);
job = ln->value;
//取走值之后,解锁
pthread_mutex_unlock(&bio_mutex[type]);
//完成出队之后,根据类型调用close、redis_fsync或者free_fn函数。
if (type == BIO_CLOSE_FILE) {
close(job->fd);
} else if (type == BIO_AOF_FSYNC) {
redis_fsync(job->fd)
} else if (type == BIO_LAZY_FREE) {
job->free_fn(job->free_args);
} else {
serverPanic("Wrong job type in bioProcessBackgroundJobs().");
}
//释放任务空间
zfree(job);
// 加锁删除队列中这个任务
pthread_mutex_lock(&bio_mutex[type]);
listDelNode(bio_jobs[type],ln);
bio_pending[type]--; //队列中pending的数量减一
//如果bioWaitStepOfType()上有阻塞的线程,则解除阻塞。
pthread_cond_broadcast(&bio_step_cond[type]);
}
}
线程类型 三种 0 关闭文件 1 aof 2 lazy free
#define BIO_CLOSE_FILE 0 /* Deferred close(2) syscall. */
#define BIO_AOF_FSYNC 1 /* Deferred AOF fsync. */
#define BIO_LAZY_FREE 2 /* Deferred objects freeing. */
//代表有上面三种类型的线程
#define BIO_NUM_OPS 3复制
上面的代码主要流程是,先判断当前的队列是否是空的,若是空的,则等待。不然,从队列中取出一个job结构,而且根据线程的类型决定调用什么函数。这里的类型是在建立线程的时候传入的参数得到的,是0 或者 1或者2。得到类型之后,从job里面取出fd做为参数,调用close函数或者redis_fsync函数。fd是一个文件描述符,因此,在任务加入队列的时候,只是把一个文件描述符入队列而已,这也就是为何bio_job结构体会设计的如此简单。但是lasy free的参数是free_args,是一个数组,注意区分。
3. Aof引用Bio
Aof 持久化是redis的两大持久化方式之一,其会以字符串的形式把对redis的每个操做都先记录在内存的一个buffer中,而后写入文件,而且在适当的时间使用fsync将数据刷入磁盘,而调用fsync的其中一种方式就是使用上面介绍的bio系统
,其使用的方式遵循了上面说的三个步骤
。
首先,在server.c中的main函数里面,有一个InitServerLast函数,其内部调用了bioInit函数,完成了bio系统的初始化,这样,相关的队列结构被创建,后台线程也被建立了。在redis主循环被启动之后,会进入持久化的时机,调用flushAppendOnlyFile函数,完成aof持久化工做。这个函数会处理aof相关的配置以及优化等各种问题,在本文只关注对bio系统的使用,其相关代码如下:
void flushAppendOnlyFile(int force) {
int sync_in_progress = 0;
...
if (server.aof_fsync == AOF_FSYNC_EVERYSEC)
sync_in_progress = aofFsyncInProgress();
...
/* Perform the fsync if needed. */
if (server.aof_fsync == AOF_FSYNC_ALWAYS) {
....
} else if ((server.aof_fsync == AOF_FSYNC_EVERYSEC &&
server.unixtime > server.aof_last_fsync)) {
if (!sync_in_progress) {
//后台执行fsync
aof_background_fsync(server.aof_fd);
server.aof_fsync_offset = server.aof_current_size;
}
server.aof_last_fsync = server.unixtime;
}
...
}
//如果一个AOf fsync当前已经在Bio后台线程中执行,返回true
int aofFsyncInProgress(void) {
return bioPendingJobsOfType(BIO_AOF_FSYNC) != 0;
}
//启动后台线程 执行aof的fsync操作
void aof_background_fsync(int fd) {
//这里就是调用Bio的创建异步任务的核心函数 发起入队操作
bioCreateFsyncJob(fd);
}复制
能够看到,其经过bioPendingJobsOfType来检查当前队列处理的状况,如果返回false代表没有Bio线程在处理,那么会调用aof_background_fsync函数,而这个函数又会调用bioCreateFsyncJob来将aof任务加入队列。因为在前面已经完成了线程的建立,在队列中有任务的时候,线程就会启动,而且经过上面讲的redis_fsync函数完成持久化操做。
4. 大Key删除用Bio(Bio新增lazy free)
大Key删除或者flushdb清空数据库等操作性能非常低下,而且会阻塞主线程。目前有一种方案就是渐进式rehash,但是Redis作者并没有考虑渐进式rehash方案,而采用在Bio中新增一个成员lazyfree。为何没有采用渐进式rehash方案呢?作者说当我们删除一个集合的时候,可能删除集合中元素的速度尚未客户端向集合中添加元素的速度快,那我们删除的工做看起来是永远也没法完成了。
为了解决以上问题, redis 4.0 引入了lazyfree的机制,它能够将删除键或数据库的操做放在后台线程里执行, 从而尽量地避免服务器阻塞。
lazyfree的原理不难想象,就是在删除对象时只是进行逻辑删除,而后把对象丢给后台,让后台线程去执行真正的删除,避免因为对象体积过大而形成阻塞。
源码:
源码太多 影响到阅读了 这里只是把流程串起来哈
使用unlink删除大key 逻辑是:
void unlinkCommand(client *c) {
delGenericCommand(c, 1);
}
delGenericCommand->dbAsyncDelete->freeObjAsync->bioCreateLazyFreeJob
看到了吧 最后调用的就是Bio系统中的bioCreateLazyFreeJob去创建任务并且入队列复制
5. 小结
能看到这里我相信您对这篇文章很感兴趣了,写这篇文章也是鼓足了很大勇气,因为要分析很多源码并且贴出来,第一得花大量时间去研究,第二贴出来害怕占用空间影响阅读,这俩对于我来说是挑战,但是对于你们读者来说只要能接受并且好好消化掉也是挑战。但是我相信只要付出,总有柳暗花明的那一天。加油。
Bio设计模式-基于锁和共享变量实现的多线程生产者消费者模型大家应该记住,这些也都是咱们在系统设计和编码中值得学习和借鉴的地方。最后,再次感谢坚持到最后的读者!
参考:
https://github.com/redis/redis/blob/c1718f9d862267bc44b2a326cdc8cb1ca5b81a39/src/lazyfree.c
https://github.com/redis/redis/blob/64f6159646337b4a3b56a400522ad4d028d55dac/src/db.c
https://github.com/redis/redis/blob/7ff7536e2c55a8a624eb52ffc35c08441425e683/src/bio.c