谱系图是层次聚类分析中一种常见形式,聚类分析是指在n维空间中将点分配到类的一种方法。本文对R语言中绘制谱系图的绘图函数及相关package进行汇总,展示不同形式的谱系图绘制过程。
1.数据准备
以国家统计局[1]官方网站上关于2020年我国31个省份人均GDP【单位:元】及年末常住人口【单位:万人】数据(未包含港澳台数据)为例,通过聚类分析构建谱系图。数据如下图所示,可在后台回复【20220114】获取。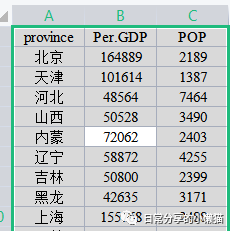
2.数据处理
前期数据处理主要包括读入数据、为数据指定行名、选择待分析的数据列、对数据进行标准化等。
setwd("C:\\Users\\Acer\\Desktop") #设置工作路径
data <- read.csv("hclust.csv") #读入数据
head(data) #查看数据前几行
# province Per.GDP POP
#1 北京 164889 2189
#2 天津 101614 1387
#3 河北 48564 7464
str(data) #查看数据结构
#'data.frame': 31 obs. of 3 variables:
# $ province: chr "北京" "天津" "河北" "山西" ...
# $ Per.GDP : int 164889 101614 48564 50528 72062 58872 50800 42635 155768 121231 ...
# $ POP : int 2189 1387 7464 3490 2403 4255 2399 3171 2488 8477 ...
rownames(data) <- data$province #为数据指定行名
data <- data[,2:3] #选择进行谱系图聚类的数据。这里选择第2~3列,去除首列,因为首列为省份名字
head(data) #查看处理后的数据
# Per.GDP POP
#北京 164889 2189
#天津 101614 1387
#河北 48564 7464
data.s <- scale(data) #对数据进行标准化处理
head(data.s) #查看标准化后的数据
# Per.GDP POP
#北京 3.00399672 -0.77270137
#天津 0.98409230 -1.03531040
#河北 -0.70940322 0.95455881复制
3.使用hclust()函数绘制谱系图
3.1 确定计算距离方法与聚类方法
通过第2部分前期数据处理,接下来使用hclust() 与dist() 函数来绘制谱系图。其中dist() 函数用来计算数据之间的距离,hclust() 函数用来确定聚类方法,基本形式为:hclust(dist(data, method = "method"), method = "method")。此外,计算距离的方法有"euclidean"(欧几里德距离), "maximum", "manhattan"(曼哈顿距离), "canberra"(堪培拉距离), "binary", "minkowski"(闵可夫斯基距离);聚类方法有"ward.D", "single", "complete", "average", "mcquitty", "median", "centroid", "ward.D2"。
hc <- hclust(dist(data.s)) #默认情况下计算距离方法为“complete”,聚类方法为“euclidean”
hc #查看聚类结果
#Call:
#hclust(d = dist(data.s))
#Cluster method : complete
#Distance : euclidean
#Number of objects: 31
hc_user <- hclust(dist(data.s, method="manhattan"), method = "ward.D") #自定义Cluster method与Distance method
hc_user #查看聚类结果
#Call:
#hclust(d = dist(data.s, method = "manhattan"), method = "ward.D")
#Cluster method : ward.D
#Distance : manhattan
#Number of objects: 31复制
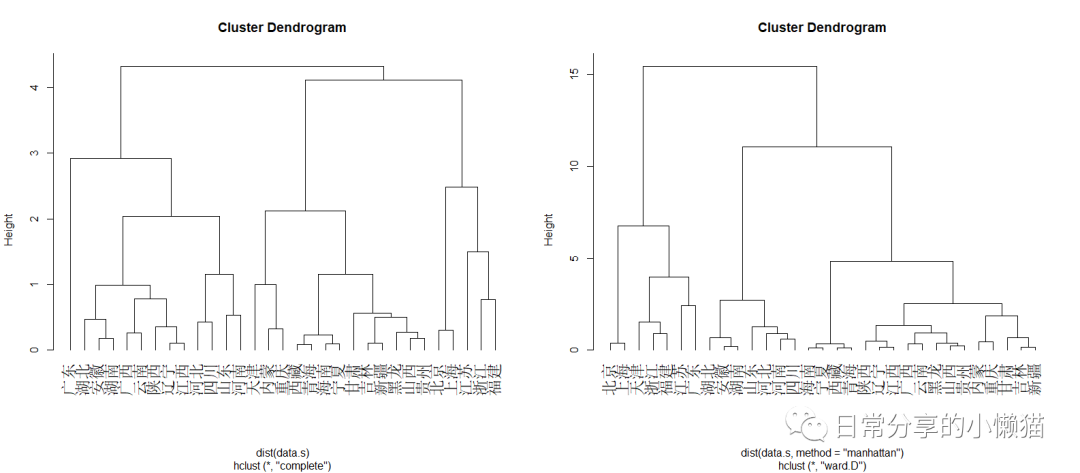
3.2 对聚类后的结果进行可视化
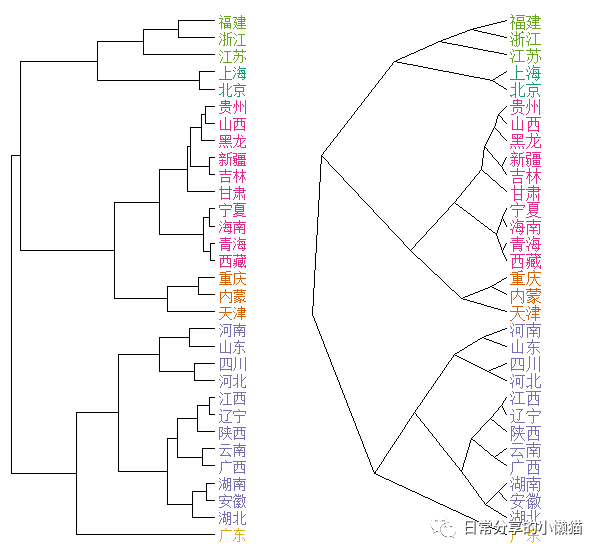
plot(hc, hang = -1) #左图,使用hang = -1可以使聚类标签对齐
plot(hc_user, hang = -1) #右图复制
4.使用ape包绘制谱系图
关于层次聚类还可以使用ape包(Analyses of Phylogenetics and Evolution)来绘制聚类图。本部分内容主要参考Gaston Sanchez[2]的Visualizing Dendrograms in R。
4.1 安装与加载package
install.packages("ape") #安装package
install.packages("RColorBrewer") #安装package
library(ape) #加载
library(RColorBrewer) #加载复制
4.2 树形聚类图
plot(as.phylo(hc), cex = 1, label.offset = 0.05, font = 1) # plot basic tree:基础谱系图(左图)
plot(as.phylo(hc), type = "cladogram", cex = 1, font = 1, label.offset = 0.05) # cladogram:分支型(右图)复制
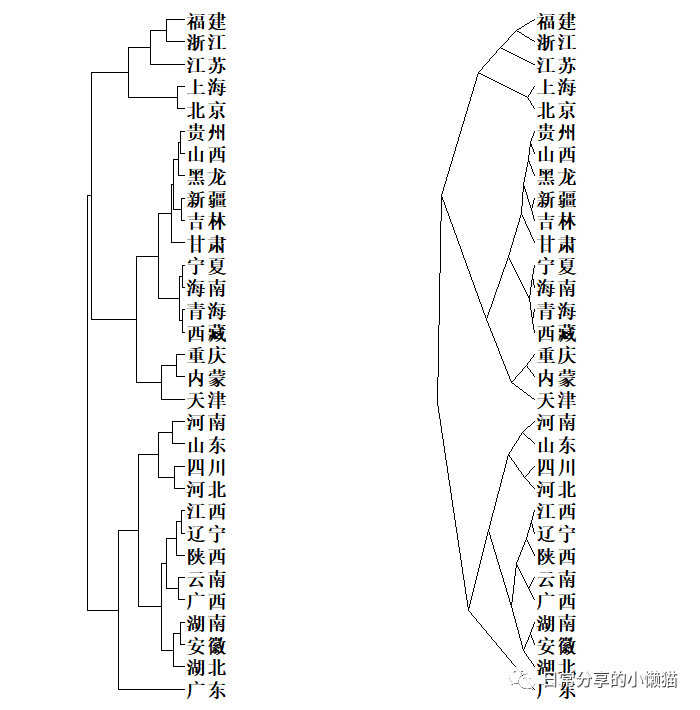
参数解读:as.phylo() 函数内为聚类后的数据,cex表示字体的大小,label.offset表示标签距离树枝的距离,font表示字体形状(粗体、斜体等),type表示选择聚类的样式,有cladogram、unrooted、fan、radial等形式。
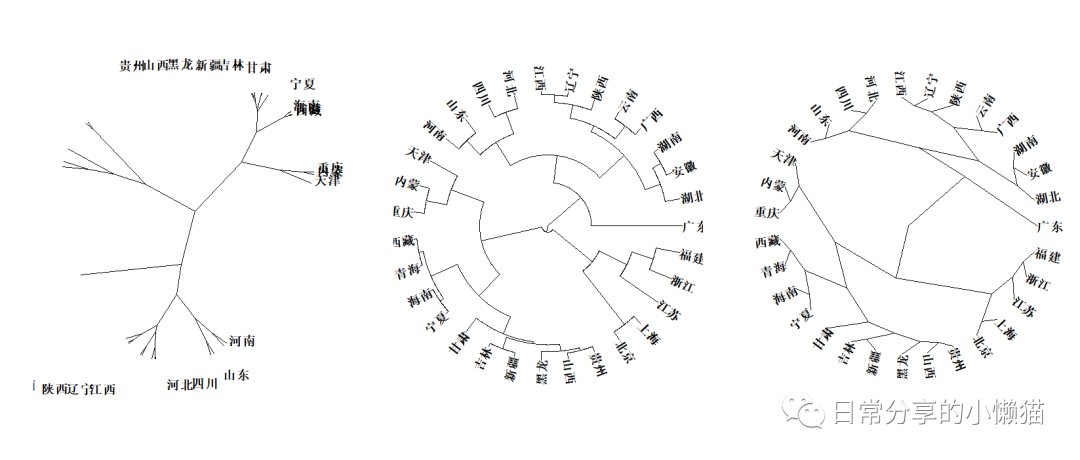
4.3 圆形聚类图
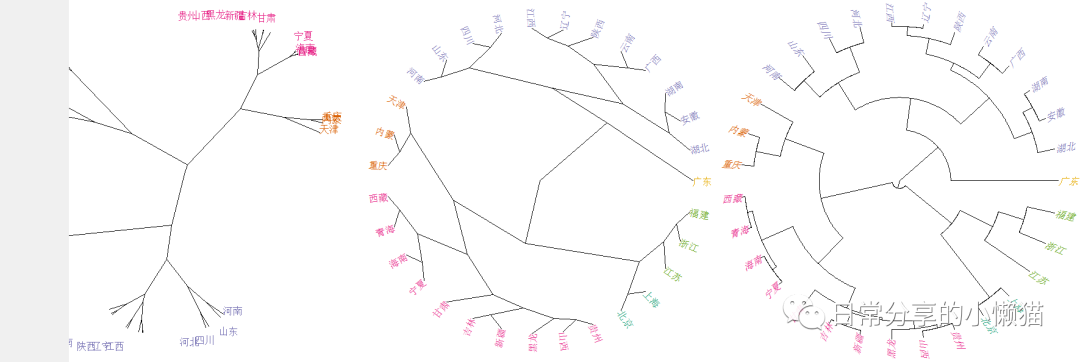
plot(as.phylo(hc), type = "unrooted",font = 2, cex = 1.5, label.offset = 0.05) # unrooted:树形(左图)
plot(as.phylo(hc), type = "fan", font = 2, cex = 1.5) # fan:扇形(中图)
plot(as.phylo(hc), type = "radial", font = 2, cex = 1.5) # radial:放射形(右图)复制
4.4 对树形聚类图进行着色(根据聚类进行着色)。主要利用tip.color() 函数,同时指定颜色的数量与聚类的数量cutree()。利用RColorBrewer包对聚类图进行着色,关于RColorBrewer包的使用可参考之前推文,见R语言绘图|如何调用RColorBrewer包对图形颜色进行修改
#设置颜色与分支(颜色数量与分支数量要一致)
mypal = brewer.pal(6, "Dark2") #设置颜色
clus6 = cutree(hc, 6) #设置分支
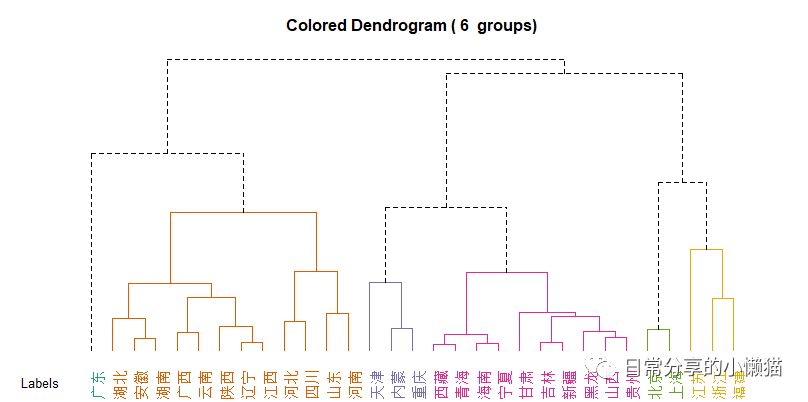
plot(as.phylo(hc), cex = 0.9, tip.color = mypal[clus6], label.offset = 0.05,font = 1, no.margin = TRUE) # plot basic tree:基础谱系图
plot(as.phylo(hc), type = "cladogram", tip.color = mypal[clus6], cex = 1, font = 1, label.offset = 0.05) # cladogram:分支形复制
4.5 对圆形聚类图进行着色(根据聚类进行着色)
plot(as.phylo(hc), type = "unrooted", tip.color = mypal[clus6], font = 1, cex = 1.5) # fan:树形
plot(as.phylo(hc), type = "radial", tip.color = mypal[clus6], font = 1, cex = 1.5) # radial:放射形
plot(as.phylo(hc), type = "fan", tip.color = mypal[clus6], label.offset = 0.01, col = "red", cex = 1.5) # fan:扇形复制
5.使用A2R包绘制谱系图
A2R包未发布在CRAN上,需要以外部形式进行加载,关于A2R包的使用可以进一步参考A2Rplot[3]中的介绍。
source("http://addictedtor.free.fr/packages/A2R/lastVersion/R/code.R") #加载A2R包
cols = brewer.pal(6, "Dark2") #设置颜色
A2Rplot(hc, k = 6, boxes = FALSE, col.up = "black", col.down = cols, lty.up= 2, lwd.up = 1, lty.down = 1, lwd.down = 1)复制
参数解读:hc为聚类后的数据,k为分类数量,boxes为FALSE时表示不加边框,boxes为TRUE时表示加边框,col.up与col.down分别为上、下线条颜色,lty为线条类型,lwd为线条厚度。
A2Rplot(hc, k = 6, boxes = TRUE, col.up = "black", col.down = cols, lty.up= 2, lwd.up = 1, lty.down = 1, lwd.down = 1)复制

6.输出谱系图数据
cluserdata <- cutree(hc, k=6) #hc为hclust()聚类后的data,k用来指定聚类的类别
#北京 天津 河北 山西 内蒙 辽宁 吉林 黑龙 上海 江苏 浙江 安徽 福建 江西 山东 河南 湖北 湖南 广东 广西 海南 重庆 四川 贵州 云南
# 1 2 3 4 2 3 4 4 1 5 5 3 5 3 3 3 3 3 6 3 4 2 3 4 3
#西藏 陕西 甘肃 青海 宁夏 新疆
# 4 3 4 4 4 4
write.csv(cluserdata, file = "cluster.csv") #写入csv文件复制
7.其他
plot() 函数是R语言绘图中一个常见的基础绘图函数,具体解读可参考R语言plot函数参数合集[4]。其基本参数如下:
#plot()绘图函数基础参数
plot(x, y = NULL, type = "p", xlim = NULL, ylim = NULL,
log = "", main = NULL, sub = NULL, xlab = NULL, ylab = NULL,
ann = par("ann"), axes = TRUE, frame.plot = axes,
panel.first = NULL, panel.last = NULL, asp = NA,
xgap.axis = NA, ygap.axis = NA,
...)复制
如有帮助请多多点赞哦!
参考资料
国家统计局: https://data.stats.gov.cn/index.htm
[2]Gaston Sanchez. Visualizing Dendrograms in R: https://rstudio-pubs-static.s3.amazonaws.com/1876_df0bf890dd54461f98719b461d987c3d.html
[3]A2Rplot.hclust {A2R}: http://addictedtor.free.fr/packages/A2R/lastVersion/html/A2Rplot.hclust.html
[4]R语言plot函数参数合集: https://blog.csdn.net/glodon_mr_chen/article/details/79830576









